研究人员开发了破译拼接中位置规则的方法
由中国科学院生物物理研究所薛元潮教授领导的研究团队开发了一种与特定RNA结合蛋白(RBP)相关的原位RNA-RNA接触的全球分析新方法,并揭示了PTBP1相关RNA环调节盒式外显子剪接的位置机制。
这项研究于22月<>日在线发表在《分子细胞》上。
在真核生物中,相同的前mRNA可以产生多种蛋白质亚型,通过交替剪接执行相似或不同的生物学功能。一些长期存在的模型提出,RBP可以通过调节长距离RNA-RNA相互作用(RRI)来调节选择性剪接。然而,缺乏直接的实验证据。
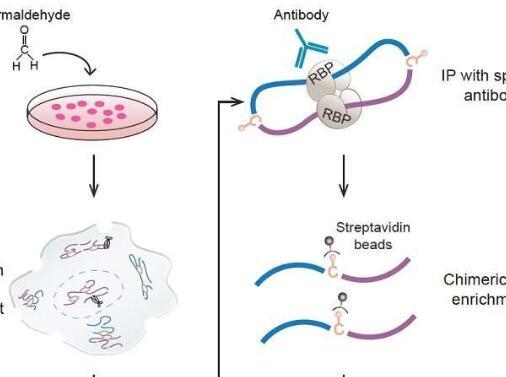
为了填补知识空白,研究人员创建了一种捕获RIC-seq(CRIC-seq)方法,通过利用RIC-seq技术和免疫沉淀介导的富集原理来绘制特定的RBP相关RRI。RIC-seq 技术由薛教授的团队于 2020 年开发,用于对所有表达 RBP 介导的 RRI 进行全球分析,而不是对特定的 RRI 进行全球分析。
利用CRIC-seq方法和自主研发的数据分析管道,研究人员获得了PTBP1、hnRNPA1或SRSF1介导的高置信度RRIs,并生成了功能性3D RNA图谱,从RNA空间构象的新角度研究这些RBP的位置机制。
与hnRNPA1类似,但与SRSF1不同,研究人员发现,当增强盒式外显子的剪接时,PTBP1介导的RNA环倾向于富集在内含子的一侧。相反,当抑制剪接时,PTBP1介导的RNA环倾向于跨越盒式外显子。这些发现表明,PTBP1可以通过以位置依赖性方式介导特定的RNA环来调节盒式外显子剪接。
根据基于微型基因的分析,研究人员发现,只有野生型(WT)PTBP1,而不是C23S突变型PTBP1,可以形成同源二聚体来挽救PTBP1敲低后相应的剪接变化。
此外,CRISPR-dCasRx介导的系留测定证实,由WT PTBP1设计的RNA环可以调节非PTBP1调节的盒式外显子的使用。这些结果表明,PTBP1二聚化可以驱动RNA环来调节剪接。
此外,通过将癌症相关的剪接数量性状位点(sQTL)与PTBP1相关的RRI相结合,研究人员确定了121个通过改变RNA环可能影响剪接的sQTL。出乎意料的是,位于HERPUD2内含子环中的一个sQTL可以诱导外显子7跳跃,从而产生一个短的亚型来促进HeLa细胞的增殖。这些结果证明了癌症相关的非编码突变如何调节RNA空间构象以促进肿瘤细胞生长。
免责声明:本文由用户上传,与本网站立场无关。财经信息仅供读者参考,并不构成投资建议。投资者据此操作,风险自担。 如有侵权请联系删除!
-
【经典句子大全】在日常生活中,无论是写作、演讲还是表达情感,一句经典的句子往往能起到画龙点睛的作用。它...浏览全文>>
-
【经典句句戳心的话】在生活的长河中,有些话虽然简单,却能直击人心,让人久久不能忘怀。这些句子往往蕴含着...浏览全文>>
-
【经典精辟句子】在日常生活中,我们常常会遇到一些让人深思、令人难忘的句子。这些句子简洁有力,富有哲理,...浏览全文>>
-
【经典惊悚片有哪些】惊悚片以其紧张的节奏、扣人心弦的情节和令人不安的氛围,成为电影史上最受欢迎的类型之...浏览全文>>
-
【经典教师简单自我介绍】在教育工作中,教师的自我介绍是展示个人风格、教学理念和职业态度的重要方式。一个...浏览全文>>
-
【经典简短句子一句话】在日常生活中,一句经典而简短的句子往往能给人留下深刻印象。它们简洁有力,蕴含深意...浏览全文>>
-
【禁不住念什么】在日常生活中,我们常常会遇到一些词语让人“禁不住”去读、去想,甚至去写。其中,“禁不住...浏览全文>>
-
【禁闭岛结局】《禁闭岛》(英文名:Shutter Island)是一部由马丁·斯科塞斯执导,莱昂纳多·迪卡普里奥主演...浏览全文>>
-
【禁闭岛的结局真相】《禁闭岛》(英文名:Shutter Island)是一部由马丁·斯科塞斯执导,莱昂纳多·迪卡普里...浏览全文>>
-
【靳字怎么读】在日常生活中,我们经常会遇到一些不常见的汉字,这些字不仅发音复杂,含义也常常让人感到困惑...浏览全文>>
